معرفی سایتها و پایگاههای آنلاین ساخت درخت فیلوژنی
مقدمهای بر پایگاهها و وبسایتهای آنلاین برای ساخت درخت فیلوژنتیک
تحلیل فیلوژنتیک در قلب زیستشناسی تکاملی مدرن قرار دارد. مطالعه روابط تکاملی میان سازوارهها (organisms)، ژنها و ژنومها از ردهبندیهای توصیفی مبتنی بر مورفولوژی (ریختشناسی) به سمت چارچوبهای محاسباتی پیشرفتهای تحول یافته است که میتوانند میلیونها کاراکتر را در هزاران تاکسون پردازش کنند. در مرکز این تحول، درخت فیلوژنتیک (phylogenetic tree) قرار دارد؛ مدلی گرافیکی که فرضیههایی درباره نیاکان و تبارزایی ارائه میدهد.
برای دههها، ساخت چنین درختهایی نیازمند آموزش تخصصی، دسترسی به رایانههای قدرتمند، و دانش پیشرفته برنامهنویسی و آمار بود. اما در سالهای اخیر، ظهور ابزارهای وبمحور و پایگاههای داده آنلاین دسترسی به روشهای فیلوژنتیک را دموکراتیک کرده است. اکنون پژوهشگران، مدرسان و حتی دانشجویان میتوانند بهراحتی درختهای تکاملی را بسازند و تحلیل کنند. این تغییر نهتنها سرعت کشفیات علمی را افزایش داده، بلکه مشارکت جهانی در مطالعات فیلوژنتیک را به کلاسها و آزمایشگاههای سراسر دنیا گسترش داده است.
رشد دادهها و نیاز به تحلیل فیلوژنتیک
با گسترش دادههای زیستی، نیاز به استنتاج فیلوژنتیک (phylogenetic inference) به شدت افزایش یافته است. فناوریهای توالییابی پرظرفیت (High-throughput sequencing) امکان توالییابی کامل ژنومها را تنها در چند ساعت فراهم کردهاند و حجم عظیمی از دادههای مولکولی تولید میکنند.
در کنار این، پایگاههای داده آنلاین مانند GenBank، Ensembl و UniProt منابع بیسابقهای برای دسترسی به توالیها در سراسر موجودات فراهم کردهاند. اکنون چالش اصلی، کمبود داده نیست، بلکه توانایی در سازماندهی، همترازسازی (alignment) و تحلیل دادهها برای ساخت فرضیههای تکاملی معتبر است.
ابزارهای فیلوژنتیک آنلاین این مشکل را حل میکنند، زیرا بستری کاربرپسند ارائه میدهند که در آن میتوان بازیابی دادهها، همترازسازی توالیها، ساخت درخت و مصورسازی نتایج را بدون نیاز به زیرساخت محاسباتی محلی انجام داد.
دسترسی و دموکراتیکسازی ابزارهای فیلوژنتیک
یکی از بزرگترین تأثیرات ابزارهای آنلاین فیلوژنتیک، افزایش دسترسی برای پژوهشگران در محیطهای متنوع است. در گذشته، اجرای تحلیلهایی مانند حداکثر درستنمایی (Maximum Likelihood) یا استنباط بیزی (Bayesian Inference) نیازمند ابررایانهها یا خوشههای محاسباتی بود.
امروز، بسیاری از این الگوریتمها از طریق سرورهای ابری (cloud-based servers) در دسترس هستند، جایی که بار محاسباتی از راه دور مدیریت میشود. این موضوع امکان مشارکت پژوهشگران در مناطق در حال توسعه یا مؤسسات با منابع محدود را فراهم کرده است.
همچنین، بسیاری از سرورها دارای رابطهای تعاملی مصورسازی (visualization interfaces) هستند که امکان بررسی درخت، حاشیهنویسی (annotation) و جزئیات شاخهها را بهصورت پویا فراهم میکنند. این تعاملی بودن، تفسیر درختها را آسانتر و تولید فرضیههای جدید را تشویق میکند.
ابزارهای آنلاین و تنوع آنها
رشد بیوانفورماتیک (bioinformatics) به تنوع این پلتفرمهای آنلاین کمک کرده است. برای مثال:
-
MEGA Online، IQ-TREE Web و PhyML Web Server ابزارهایی برای ساخت درخت با استفاده از الگوریتمها و مدلهای جانشینی مختلف ارائه میدهند.
-
iTOL (Interactive Tree of Life) یک پلتفرم مصورسازی است که امکان حاشیهنویسی غنی فراهم میکند، مانند نگاشت ویژگیها، دادههای بیان ژن، یا اطلاعات اپیدمیولوژیک روی فیلوژنیها.
این ترکیب دادهها، درخت فیلوژنتیک را از یک نمودار ایستا به یک منبع پویا و چندبعدی برای استنباط زیستی تبدیل میکند.
نمونه مهم، اپیدمیولوژی است، جایی که درختهای فیلوژنتیک برای ردیابی خاستگاه و انتشار پاتوژنهای ویروسی مانند آنفلوآنزا یا SARS-CoV-2 استفاده میشوند. پلتفرم Nextstrain یک مثال بارز است که نظارت فیلوژنتیک بلادرنگ بر تکامل جهانی ویروسها را امکانپذیر کرده است.
ارزش آموزشی ابزارهای فیلوژنتیک آنلاین
ارزش آموزشی این ابزارها نیز بسیار زیاد است. در کلاسهای درس، مدرسان میتوانند مفاهیم بنیادی تکامل، زیستشناسی مولکولی و سیستماتیک را با استفاده از تمرینهای عملی و دادههای واقعی آموزش دهند.
به جای نمودارهای انتزاعی، دانشجویان میتوانند توالیهای ژنی را از پایگاههای عمومی دانلود کنند، آنها را همترازسازی کنند و درختهایی بسازند که تاریخچههای واقعی تکاملی را نشان میدهد.
این یادگیری تجربی فرآیند علمی را تقویت میکند و اهمیت بیوانفورماتیک را در زیستشناسی برجسته میسازد. علاوه بر این، از آنجا که بسیاری از ابزارهای آنلاین دارای رابطهای گرافیکی ساده و شهودی هستند، یادگیری آسانتر میشود و حتی دانشجویانی بدون پسزمینه برنامهنویسی میتوانند در تحلیل فیلوژنتیک مشارکت کنند.
محدودیتهای ابزارهای آنلاین
ابزارهای فیلوژنتیک آنلاین در کنار مزایا، محدودیتهایی نیز دارند:
-
محدودیت در اندازه دادهها و شدت محاسبات
-
محدودیت در گزینههای مدلهای تکاملی
-
تکیه بر محاسبات از راه دور، که نیازمند اعتماد به پیادهسازی الگوریتمها است
این محدودیتها برای مدیریت ظرفیت سرورها در برابر تقاضای کاربران طراحی شدهاند، اما ممکن است در تحلیلهای بزرگ یا پیچیده کاربردپذیری را کاهش دهند.
با این وجود، برای بسیاری از کاربران، بهویژه تازهواردان به این حوزه، این پلتفرمها نقطه شروع ضروری برای ورود به تحلیل فیلوژنتیک هستند.
نقش پایگاههای داده در فیلوژنی آنلاین
گسترش پایگاههای دادهای که این ابزارها را تغذیه میکنند، بخش کلیدی از اکوسیستم فیلوژنتیک آنلاین است.
-
GenBank (توسط NCBI): بزرگترین مخزن توالیهای نوکلئوتیدی از موجودات مختلف.
-
Ensembl: دادههای ژنومی در مقیاس کامل.
-
UniProt: اطلاعات دقیق پروتئینها.
این پایگاهها تنها مخزن داده نیستند، بلکه ابزارهای جستوجو و بازیابی نیز ارائه میدهند که به کاربران امکان میدهد مجموعه دادههای خود را سریعاً گردآوری کنند. اتصال مستقیم آنها به ابزارهای فیلوژنتیک، فرآیند حرکت از داده خام به تفسیر تکاملی را بسیار ساده میکند.
پژوهش بلادرنگ و همکاری آنلاین
بُعد دیگری از ابزارهای فیلوژنتیک آنلاین، حرکت به سمت پژوهش بلادرنگ و همکاری گروهی است.
-
iTOL به کاربران اجازه میدهد درختهای حاشیهنویسیشده را با همکاران خود به اشتراک بگذارند.
-
Nextstrain بر پایه اشتراکگذاری دادهها ساخته شده است و بازسازیهای فیلوژنتیک بهروز ویروسهای بیماریزا را فراهم میکند.
این نوع همکاری تا یک دهه پیش غیرقابل تصور بود، اما اکنون به استانداردی در اپیدمیولوژی و بومشناسی میکروبی تبدیل شده است.
استانداردسازی و تکرارپذیری
استفاده از ابزارهای آنلاین پرسشهای مهمی درباره استانداردسازی و بازتولیدپذیری (reproducibility) مطرح میکند.
-
پلتفرمهای مختلف ممکن است پیادهسازی الگوریتمها را کمی متفاوت انجام دهند.
-
برخی رابطها جزئیات انتخاب پارامترها را پنهان میکنند، که میتواند تفسیر نتایج را دشوار سازد.
به همین دلیل، کاربران باید دانش پایهای درباره روشهای فیلوژنتیک داشته باشند، حتی اگر به پلتفرمهای خودکار متکی باشند. بنابراین، منابع آموزشی، راهنماها و مستندات کاربری نقش مهمی در تضمین اعتبار و قابلیت تفسیر نتایج دارند.
۲. مبانی درختهای فیلوژنتیک
درختهای فیلوژنتیک سنگبنای زیستشناسی تکاملی هستند و چارچوبی ساختاری برای نمایش فرضیهها درباره روابط میان موجودات، ژنها یا گروههای ردهبندی بالاتر فراهم میکنند. این درختها صرفاً نمودارهای انتزاعی نیستند، بلکه مدلهای علمی هستند که شواهد حاصل از توالیهای مولکولی، صفات ریختشناسی یا دیگر ویژگیهای زیستی را خلاصه میکنند.
بنابراین، درک اصول پایهای درختهای فیلوژنتیک برای هر کسی که میخواهد آنها را تفسیر یا بسازد ضروری است. هرچند ابزارهای محاسباتی مدرن بیشتر فرآیند ساخت درخت را خودکار کردهاند، تفسیر این درختها نیازمند دقت در ساختار، فرضیات زیربنایی و روششناسی بهکاررفته در آنهاست. در غیر این صورت، حتی پیشرفتهترین ابزارهای آنلاین میتوانند به برداشتهای نادرست منجر شوند.
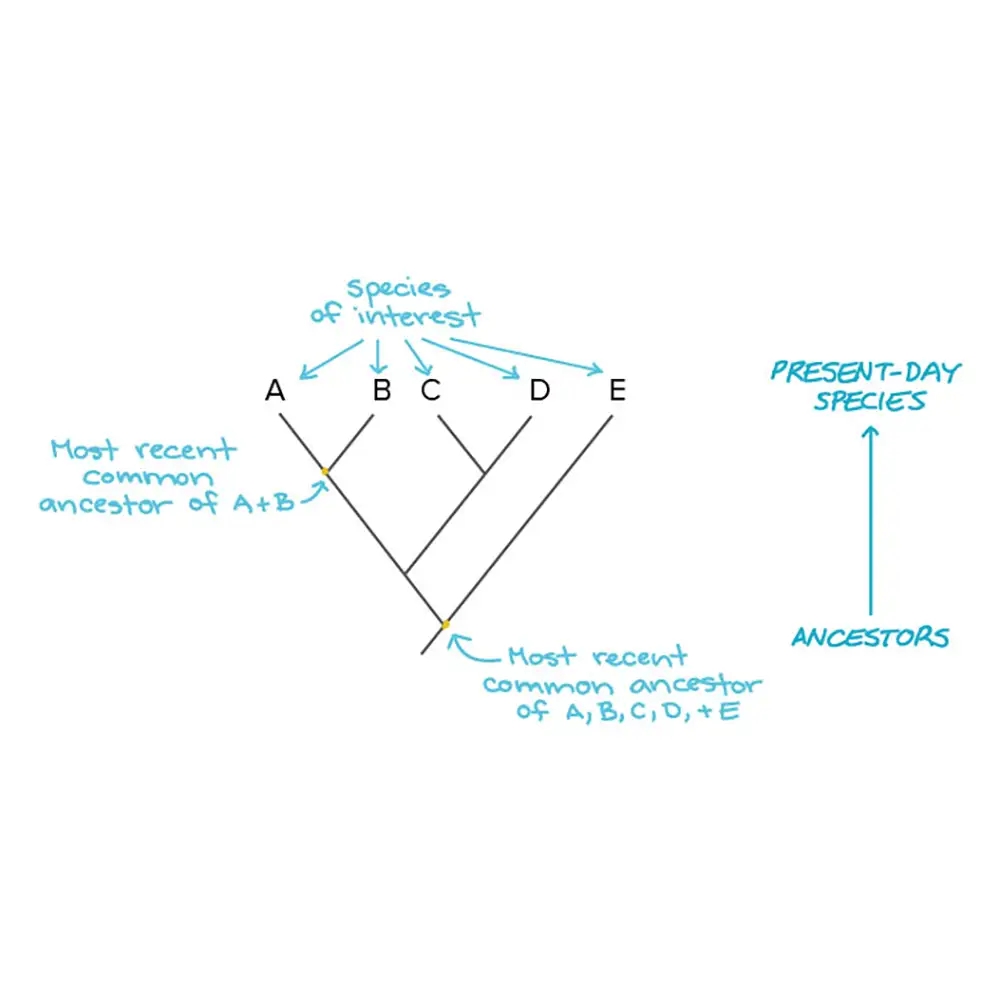
ساختار پایهای درخت فیلوژنتیک
در سادهترین سطح، درخت فیلوژنتیک یک نمودار شاخهای است که مسیرهای تکاملی بین موجودات یا توالیهای ژنتیکی را نمایش میدهد.
-
گرهها (nodes): نقاطی که شاخهها از هم جدا میشوند و نیاکان مشترک فرضی را نشان میدهند.
-
انتهای شاخهها (leaves/tips): تاکسونهایی که مورد مطالعهاند (گونهها، سویهها، ژنها یا پروتئینها).
-
شاخهها (branches): نمایانگر تبارهای تکاملی (lineages) هستند و الگوی اتصال آنها فرضیهای درباره ترتیب رخداد انشعابها ارائه میدهد.
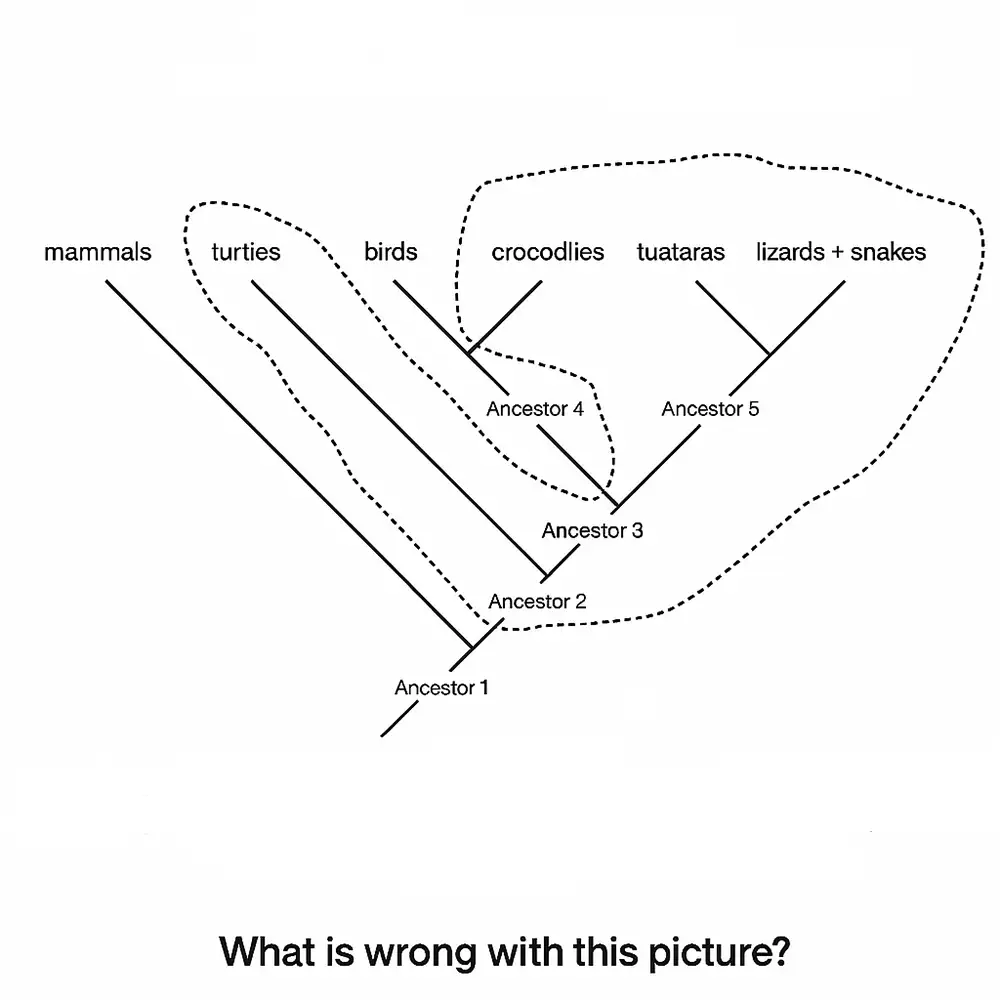
نکته مهم: درختهای فیلوژنتیک نشاندهنده تبارزایی مشترک هستند، نه رابطه مستقیم والد–فرزندی. مثلاً وقتی درختی نشان میدهد که انسان و شامپانزه یک نیاکان مشترک داشتهاند، به این معنا نیست که یکی مستقیماً از دیگری تکامل یافته است، بلکه هر دو از یک تبار منقرضشده منشعب شدهاند.
درختهای ریشهدار و بیریشه
-
درخت ریشهدار (rooted tree): دارای ریشهای مشخص است که نمایانگر جد مشترک همه تاکسونها در درخت میباشد. این ریشه جهت زمانی تکامل را نشان میدهد. چنین درختهایی برای استنتاج تکاملی ضروری هستند (مثلاً تعیین اینکه کدام شاخه قدیمیتر است).
-
درخت بیریشه (unrooted tree): تنها روابط میان تاکسونها را نشان میدهد، بدون اشاره به نیاکان مشترک یا جهت تکامل. این نوع درخت توان استنباطی محدودتری دارد.
در عمل، پژوهشگران از برونگروهها (outgroups) – تاکسونهایی که نسبت به گروه مورد مطالعه دورتر هستند – برای ریشهگذاری درخت و تعیین جهت تکامل استفاده میکنند.
کلا دوگرام، فیلوگرام و کرونوگرام
-
کلا دوگرام (cladogram): فقط الگوی شاخهبندی را نشان میدهد و اطلاعی از میزان تغییر تکاملی نمیدهد.
-
فیلوگرام (phylogram): علاوه بر الگوی شاخهبندی، طول شاخهها را متناسب با میزان تغییر ژنتیکی یا فاصله تکاملی نشان میدهد.
-
کرونوگرام (chronogram): طول شاخهها را به زمان مطلق کالیبره میکند (معمولاً بر اساس فسیلها یا ساعتهای مولکولی).
این تمایزها اهمیت دارند، زیرا نوع درخت بر نتیجهگیریهای زیستی تأثیر میگذارد. مثلاً: یک کلا دوگرام نشان میدهد دو شاخه نزدیکاند، اما نمیتواند بگوید آیا یکی سریعتر تکامل یافته است یا نه.
دادهها در فیلوژنتیک
امروزه، توالیهای مولکولی (DNA، RNA و پروتئینها) پرکاربردترین دادهها برای ساخت درختها هستند.
-
دادههای ریختشناسی، رفتاری یا غیرمولکولی هنوز در برخی موارد (مثلاً نبود داده مولکولی) بهکار میروند، اما بیشتر مستعد مشکلاتی مثل تکامل همگرا (convergent evolution) یا انعطافپذیری فنوتیپی (phenotypic plasticity) هستند.
-
دادههای مولکولی تعداد زیادی کاراکتر قابل مقایسه فراهم میکنند و کمتر تحت تأثیر محیط قرار دارند.
به همین دلیل، با گسترش فناوریهای توالییابی و پایگاههای داده عمومی، فیلوژنتیک مولکولی به رویکرد غالب تبدیل شده است.
روشهای استنتاج درخت فیلوژنتیک
چند چارچوب اصلی وجود دارد:
-
روشهای فاصلهمحور (distance-based): مثل neighbor-joining، که بر اساس مقایسههای جفتی شباهت توالی ساخته میشوند.
-
مزیت: سریع و مناسب برای مجموعهدادههای بزرگ.
-
عیب: سادهسازی بیش از حد فرآیندهای تکاملی.
-
-
روشهای کاراکتر-محور (character-based):
-
حداکثر صرفهجویی (maximum parsimony): سادهترین درخت با حداقل تغییرات تکاملی.
-
حداکثر درستنمایی (maximum likelihood): استفاده از مدلهای آماری برای یافتن درختی که دادهها را بیشترین احتمال توضیح میدهد.
-
استنباط بیزی (Bayesian inference): توسعهیافته از درستنمایی، که احتمالات پیشین (priors) را هم در نظر میگیرد و عدمقطعیت را بهطور احتمالاتی مدیریت میکند.
-
انتخاب روش وابسته به نوع داده، سؤال پژوهش و منابع محاسباتی است.
همترازسازی توالیها (Sequence Alignment)
پیش از ساخت درخت، توالیها باید همتراز (aligned) شوند تا موقعیتهای همولوگ مقایسه شوند.
-
همترازسازی نادرست میتواند به استنتاجهای غلط منجر شود.
-
ابزارهای متداول: Clustal Omega، MUSCLE، MAFFT
-
بسیاری از پلتفرمهای آنلاین این ابزارها را مستقیماً ادغام کردهاند.
کیفیت همترازسازی تأثیر مستقیم بر دقت درخت دارد. در موارد دشوار (مثل توالیهای واگرا یا نواحی غیرکدکننده)، ممکن است نیاز به اصلاح دستی یا رویکردهای تکرارشونده باشد.
انتخاب مدل (Model Selection)
تکامل مولکولی یکنواخت نیست. مدلهای جانشینی (substitution models) برای توضیح الگوهای تغییر استفاده میشوند:
-
برخی مدلها همه تغییرات را برابر میدانند.
-
برخی دیگر تفاوت ترانزیشن/ترانسورژن، استفاده از کدون یا ناهمگنی نرخها بین جایگاهها را در نظر میگیرند.
ابزارها: jModelTest یا ModelFinder (در IQ-TREE)
-
انتخاب مدل مناسب اثر چشمگیری بر نتیجه دارد.
-
مدل نادرست → نتایج گمراهکننده
بنابراین، دقت آماری در هر مرحله—even در ابزارهای آنلاین—ضروری است.
معیارهای اطمینان (Confidence Measures)
همه شاخهها به یک اندازه مطمئن نیستند.
-
آنالیز بوتاسترپ (bootstrap analysis): بازنمونهگیری دادهها و بررسی تکرار شاخهها.
-
مقدار بالا → حمایت قوی
-
مقدار پایین → عدم قطعیت
-
-
روشهای بیزی: احتمال پسین (posterior probabilities)
پلتفرمهای آنلاین این مقادیر را معمولاً بهصورت درصد یا نمره احتمالی روی درخت نشان میدهند.
تفسیر درست این مقادیر برای جلوگیری از اعتماد بیش از حد به شاخههای ضعیف مهم است.
مصورسازی (Visualization)
مصورسازی درختها حیاتی است، نه صرفاً زیباییشناختی.
-
درختهای بزرگ (با صدها یا هزاران تاکسون) پیچیده میشوند.
-
ابزارهای خوب اجازه میدهند:
-
فشردهسازی شاخهها (collapse clades)
-
برجستهسازی تبارهای خاص
-
افزودن دادههای متادیتا (مثل موقعیت جغرافیایی یا عملکرد ژن)
-
مثال: iTOL (Interactive Tree of Life) → محیط تعاملی و قابل اشتراکگذاری.
کاربردهای عملی
درختهای فیلوژنتیک فقط برای زیستشناسی نظری نیستند:
-
ردهبندی (taxonomy): چارچوبی مبتنی بر شواهد برای طبقهبندی.
-
زیستشناسی حفاظت (conservation biology): شناسایی شاخههای متمایز تکاملی برای حفاظت.
-
پزشکی: ردیابی منشأ و انتشار پاتوژنها (مثلاً ویروسها).
-
کشاورزی: شناسایی روابط ژنتیکی میان گونههای زراعی برای اصلاح نباتات.
۳. منابع داده برای تحلیل فیلوژنتیک
ساخت درختهای فیلوژنتیک بهطور بنیادی وابسته به دسترسپذیری و کیفیت دادهها است. یک درخت تنها به اندازهی توالیها یا صفاتی که از آنها استنتاج شده قابل اعتماد است، و در عصر مولکولی، این معمولاً به معنای توالیهای DNA، RNA یا پروتئین است که از پایگاههای داده معتبر بهدست میآیند.
در چند دهه گذشته، یک زیرساخت جهانی عظیم از مخازن آنلاین ایجاد شده است تا دادههای توالی تولیدشده توسط هزاران آزمایشگاه در سراسر جهان را ذخیره، سازماندهی و منتشر کند. این مخازن رویکرد دانشمندان به تحقیقات فیلوژنتیک را دگرگون کردهاند؛ بهجای دادههای پراکنده و بایگانیهای محلی، اکنون پلتفرمهای مشارکتی و دسترسی آزاد وجود دارند که کل جامعه علمی را پشتیبانی میکنند.
برای پژوهشگرانی که علاقهمند به ساخت درخت فیلوژنتیک بهصورت آنلاین هستند، شناخت ماهیت این پایگاهها، ساختار آنها، نقاط قوت و محدودیتهایشان حیاتی است. در این بخش مهمترین منابع داده برای تحلیل فیلوژنتیک معرفی میشوند، با تمرکز بر پایگاههای بینالمللی توالیها و مخازن تخصصی که به ابزارهای فیلوژنتیک آنلاین داده میرسانند.
GenBank
یکی از برجستهترین و پرکاربردترین پایگاههای توالی، GenBank است که توسط مرکز ملی اطلاعات بیوتکنولوژی (NCBI) در ایالات متحده میزبانی میشود.
-
تأسیس: اوایل دهه ۱۹۸۰
-
محتوا: میلیاردها توالی نوکلئوتیدی از هزاران گونه در سراسر درخت زندگی
-
بهروزرسانی: روزانه برای افزودن توالیهای جدید
مزیتها برای فیلوژنتیک:
-
ارائهی توالی خام + فرادادهها (metadata) مانند ردهبندی گونه، منابع مقالات و حاشیهنویسی توالیها.
-
ادغام با ابزارهایی مثل BLAST و NCBI Taxonomy Browser که امکان شناسایی توالیهای همولوگ و آمادهسازی آنها برای همترازی و ساخت درخت را فراهم میکنند.
-
سیاست دسترسی آزاد GenBank باعث میشود پژوهشگران در سراسر جهان بدون توجه به وابستگی سازمانی، بتوانند از آن استفاده کنند.
GenBank یک سنگبنای جهانی برای تحقیقات فیلوژنتیک است.
Ensembl
Ensembl یک پروژه مشترک از مؤسسه بیوانفورماتیک اروپا (EBI) و مؤسسه ولکام تراست سانگر است.
-
تمرکز: برخلاف GenBank که جامع است، Ensembl برای حاشیهنویسی ژنوم مهرهداران و برخی ارگانیسمهای مدل طراحی شده است.
-
دادهها: شامل توالیهای ژنومی همراه با پیشبینی ژنها، ویژگیهای تنظیمی و دادههای واریاسیون.
مزیتها برای فیلوژنتیک:
-
ابزارهای ژنتیک مقایسهای (comparative genomics) → بازیابی ژنهای ارتولوگ و پارالوگ در چندین گونه.
-
رابط BioMart و دسترسی برنامهنویسی (APIs) → ساخت مجموعهدادههای بزرگ و سفارشی.
-
Ensembl Genomes → گسترش به گونههای غیرمهرهدار مثل گیاهان، قارچها، پروتیستها و باکتریها.
Ensembl فراتر از توالیهاست: روابط ژنی ساماندهیشده را هم ارائه میدهد که برای مطالعه تکامل خانوادههای ژنی بسیار ارزشمند است.
DDBJ و INSDC
بانک داده DNA ژاپن (DDBJ) همراه با GenBank و آرشیو نوکلئوتیدی اروپا (ENA) بخشی از همکاری بینالمللی پایگاه توالی نوکلئوتیدی (INSDC) را تشکیل میدهند.
-
این همکاری باعث میشود دادههایی که به هر یک از این مخازن سپرده میشوند، بهصورت روزانه بین همه همگام شوند.
-
نتیجه: یک بایگانی جهانی و هماهنگ بدون سوگیری منطقهای.
DDBJ توسط مؤسسه ملی ژنتیک ژاپن میزبانی میشود و خدمات ویژهای برای جامعه پژوهشی آسیا ارائه میدهد، اما دادههای آن در دسترس جهانی هستند.
اهمیت INSDC: تضمین دسترسی جهانی و جامع به دادههای توالی، برای تحلیلهای فیلوژنتیک بزرگمقیاس.
UniProt
UniProt (منبع جهانی پروتئین) مهمترین پایگاه برای توالی پروتئین و اطلاعات عملکردی است.
-
مدیریت مشترک: EBI، مؤسسه بیوانفورماتیک سوئیس و مرکز اطلاعات پروتئین ایالات متحده.
-
محتوا: توالیهای پروتئینی همراه با حاشیهنویسیهای عملکردی گسترده (دامنهها، جایگاههای فعال، اصلاحات پسترجمهای، تعاملات پروتئینی).
مزیتها برای فیلوژنتیک:
-
مطالعه خانوادههای پروتئینی، واگرایی عملکردی و حفاظت تکاملی.
-
UniProtKB شامل دو بخش است:
-
Swiss-Prot: ورودیهای بازبینیشده و دستی.
-
TrEMBL: ورودیهای محاسباتی و بازبینینشده.
-
پروتئینها معمولاً آهستهتر از نوکلئوتیدها تکامل مییابند، بنابراین برای بررسی روابط تکاملی عمیقتر بسیار ارزشمند هستند.
TreeBASE
TreeBASE یک پایگاه داده تخصصی برای درختهای فیلوژنتیک و مجموعهدادههای مربوطه است.
-
برخلاف GenBank یا UniProt، که داده خام ذخیره میکنند، TreeBASE تحلیلهای فیلوژنتیک منتشرشده را میزبانی میکند.
-
کاربردها:
-
بازتحلیل و مقایسه درختها
-
متاآنالیز
-
بررسی الگوهای بزرگمقیاس تکاملی
-
-
منبعی مهم برای شفافیت، بازتولیدپذیری و اشتراک داده در فیلوژنتیک.
OrthoDB
OrthoDB پایگاهی برای ژنهای ارتولوگ در طیف گستردهای از گونههاست.
-
ارتولوگها: ژنهایی در گونههای مختلف که از یک ژن نیایی مشترک از طریق رویدادهای گونهزایی منشعب شدهاند.
-
اهمیت: شناسایی ارتولوگها یک گام کلیدی در بسیاری از تحلیلهای فیلوژنتیک است.
-
OrthoDB مجموعههای از پیش محاسبهشده ژنهای ارتولوگ ارائه میدهد → صرفهجویی در زمان پژوهشگران.
ویژه در فیلوژنتیک ژنومی (phylogenomics): جایی که درختها بر اساس چندین ژن یا کل ژنوم ساخته میشوند.
پایگاههای میکروبی: SILVA و Greengenes
برای فیلوژنتیک میکروبی، پایگاههای تخصصی rRNA بسیار مهماند:
-
SILVA: ارائه همترازسازی باکیفیت و ردهبندی برای rRNA در سراسر دامنههای حیات.
-
Greengenes: گرچه دیگر بهروزرسانی نمیشود، هنوز منبعی ارزشمند برای توالیهای 16S rRNA در مطالعات بومشناسی میکروبی است.
این پایگاهها دادههای پالایششدهای ارائه میدهند که برای ساخت فیلوژنیهای دقیق میکروبی ضروریاند.
پایگاههای ویروسی: GISAID
GISAID (ابتکار جهانی برای اشتراک دادههای آنفلوانزا) ابتدا برای ویروسهای آنفلوانزا ایجاد شد و سپس به دیگر پاتوژنها، بهویژه SARS-CoV-2 گسترش یافت.
-
در همهگیری COVID-19 نقشی محوری داشت: فراهمکردن دسترسی بلادرنگ به توالیهای ژنومی ویروس.
-
درختهای فیلوژنتیک ساختهشده با این دادهها مسیر انتشار و تکامل ویروس را رهگیری کردند.
-
تفاوت با GenBank: سیاست اشتراکگذاری ویژه که دسترسی آزاد را با حفظ حقوق مشارکتکنندگان داده متعادل میکند.
ارتباط و یکپارچگی پایگاهها
بسیاری از این پایگاهها با یکدیگر و با ابزارهای آنلاین فیلوژنتیک یکپارچه هستند:
-
توالیهای GenBank → استفاده مستقیم در NCBI Tree Viewer
-
دادههای UniProt → ادغام در تحلیل خانوادههای پروتئینی با PhyML یا IQ-TREE
-
دادههای Ensembl → خروجی در فرمتهای سازگار با ابزارهای همترازی مثل MUSCLE یا MAFFT
این همپوشانی و یکپارچگی (interoperability) از ویژگیهای مهم بیوانفورماتیک مدرن است و بازدهی تحلیلهای آنلاین فیلوژنتیک را بسیار افزایش میدهد.
۴. ابزارهای آنلاین فیلوژنی (Online Phylogeny Tools)
رشد سریع روشهای محاسباتی و منابع اینترنتی، زمینهی فیلوژنی (Phylogenetics) را از یک حوزهی تخصصی و محاسباتی پیچیده به یک فعالیت دسترسپذیر و جهانی تبدیل کرده است. در گذشته، ساخت درختهای فیلوژنتیکی نیازمند مهارتهای گستردهی برنامهنویسی، نصب نرمافزارهای پیچیده بر روی سیستمهای محلی، و دسترسی به منابع محاسباتی قدرتمند بود. امروزه بسیاری از این موانع کاهش یافته یا بهطور کامل برطرف شدهاند، به لطف ابزارهای آنلاین فیلوژنی.
این پلتفرمها رابطهای تحت وب ساده و شهودی در اختیار کاربران قرار میدهند و مراحل مختلف فرایند فیلوژنی ــ از بازیابی توالیها (Sequence Retrieval) تا همترازی (Alignment)، انتخاب مدل (Model Selection) و ساخت درخت (Tree Building) ــ را در یک محیط یکپارچه ترکیب میکنند. علاوه بر این، بسیاری از آنها امکانات پیشرفته برای بصریسازی (Visualization) و حاشیهنویسی (Annotation) فراهم میکنند.
برای دانشجویان، پژوهشگران و متخصصان، وجود این ابزارها بدین معناست که میتوانند تحلیلهای فیلوژنتیکی را سریعتر، بازتولیدپذیرتر و بدون نیاز به سختافزار تخصصی انجام دهند. اما برای استفادهی مؤثر از این منابع، لازم است که ویژگیها، نقاط قوت، محدودیتها و کاربردهای مناسب آنها بهدرستی درک شود.
MEGA Online (Molecular Evolutionary Genetics Analysis)
یکی از پرکاربردترین پلتفرمها، MEGA Online است که نسخهی تحت وب نرمافزار قدیمی و محبوب MEGA میباشد. MEGA سالها یکی از برنامههای اصلی در تحلیل فیلوژنی بوده است، بهدلیل رابط کاربری ساده، گزینههای روششناختی گسترده و ادغام مدلهای تکاملی.
نسخهی آنلاین MEGA این مزایا را حفظ کرده و دسترسی را آسانتر میکند، زیرا دیگر نیازی به دانلود یا نصب نرمافزار وجود ندارد. در MEGA Online، پژوهشگران میتوانند دادههای همتراز شده را وارد کنند، مدلهای تکاملی مناسب را انتخاب کرده و درختهایی را با روشهایی مانند Maximum Likelihood، Neighbor-Joining یا Maximum Parsimony بسازند.
یکی از نقاط قوت MEGA وجود چارچوب تست مدلها است که به کاربران امکان میدهد مدلهای جانشینی (Substitution Models) مختلف را مقایسه کرده و بهترین مدل برای دادههای خود را انتخاب کنند. این موضوع حیاتی است زیرا دقت استنباط فیلوژنتیکی بهشدت تحت تأثیر انتخاب مدل مناسب است.
همچنین، MEGA شاخصهای آماری قدرتمندی مانند Bootstrap Values ارائه میدهد تا کاربران بتوانند اعتماد به شاخههای درخت را ارزیابی کنند. با این حال، MEGA Online برای مجموعهدادههای کوچک تا متوسط ایدهآل است، ولی در مواجهه با دادههای بسیار بزرگ یا پیچیده محدودیتهایی دارد، بهدلیل نیازهای محاسباتی بالای روشهای مبتنی بر Likelihood.
IQ-TREE Web Server
یکی دیگر از پلتفرمهای پرطرفدار، IQ-TREE Web Server است که نسخهی تحت وب نرمافزار قدرتمند IQ-TREE میباشد. این ابزار بهخاطر سرعت، دقت و قابلیتهای پیشرفتهی انتخاب مدل شهرت جهانی دارد.
نسخهی وب، دسترسی آسان به این قابلیتها را فراهم میکند: کاربران میتوانند همترازهای توالی را بارگذاری کرده، از میان مدلهای جانشینی متنوع انتخاب کنند و درختها را با روش Maximum Likelihood بسازند.
ویژگی متمایز IQ-TREE، وجود ModelFinder است؛ الگوریتمی که بهطور خودکار بهترین مدل جانشینی را برای دادهها شناسایی میکند.
علاوه بر این، IQ-TREE از تکنیکهای نوآورانهای مانند Ultrafast Bootstrap Approximation و SH-aLRT Tests استفاده میکند، که امکان ارزیابی سریع حمایت آماری از شاخهها را فراهم میسازد، بدون کاهش دقت.
این ویژگیها باعث شده IQ-TREE برای مجموعهدادههای بزرگ و مطالعات فیلوژنومیکس (Phylogenomics) بسیار مناسب باشد. همچنین، این پلتفرم امکان تحلیلهای Partitioned Analysis را فراهم میکند، یعنی بخشهای مختلف داده (مثلاً جایگاههای کدونی یا خانوادههای ژنی) میتوانند مدلهای تکاملی متفاوتی داشته باشند.
PhyML Online
PhyML Online یکی دیگر از منابع مهم برای ساخت درختهای فیلوژنتیکی با استفاده از Maximum Likelihood است. نرمافزار PhyML سالها یکی از پرارجاعترین بستههای فیلوژنی بوده است، چون میان کارایی محاسباتی و استحکام روششناختی تعادل برقرار کرده است.
نسخهی آنلاین نیز همین تعادل را حفظ میکند و امکان اجرای سریع تحلیلها را برای مجموعهدادههای متوسط فراهم میسازد. کاربران میتوانند توالیهای همتراز شده را بارگذاری کنند، مدلهای مختلف جانشینی را انتخاب کرده و الگوریتمهای جستجو مانند Nearest-Neighbor Interchange (NNI) یا Subtree Pruning and Regrafting (SPR) را مشخص نمایند.
PhyML همچنین مقادیر Bootstrap Support و Likelihood Ratio Tests ارائه میدهد تا کاربران بتوانند قابلیت اطمینان درخت را بسنجند. هرچند بهاندازهی IQ-TREE پیشرفته نیست، اما بهخاطر سرعت و سادگی جریان کار همچنان محبوب است.
ابزارهای بصریسازی (Visualization Tools)
در کنار ابزارهای ساخت درخت، برخی پلتفرمها بر بصریسازی و حاشیهنویسی درختهای فیلوژنتیکی تمرکز دارند.
-
iTOL (Interactive Tree of Life):
شاید شناختهشدهترین ابزار در این حوزه باشد. iTOL میتواند درختهای بسیار بزرگ را مدیریت کرده و تصویری کاملاً قابل تنظیم ارائه دهد. کاربران میتوانند شاخهها را رنگبندی کنند، نوکها (Tips) را با اطلاعات تاکسونومیک یا عملکردی برچسبگذاری کرده و کلادها (Clades) را باز یا بسته کنند.
همچنین امکان اضافه کردن Heatmap، Bar Chart یا متادادههای خارجی وجود دارد. iTOL ابزاری ضروری در بومشناسی میکروبی، اپیدمیولوژی و سایر حوزهها است. -
NCBI Tree Viewer:
این ابزار بهطور مستقیم با GenBank و منابع NCBI یکپارچه است. کاربرانی که با BLAST توالی پیدا میکنند، میتوانند بهسرعت یک درخت Neighbor-Joining ساخته و آن را در Tree Viewer مشاهده کنند. این ابزار اگرچه بهاندازهی iTOL قابل شخصیسازی نیست، اما قدرتش در ادغام با پایگاهدادهی عظیم NCBI است.
ابزارهای تخصصی برای میکروبها و ویروسها
-
PhyloPhlAn:
پلتفرمی تخصصی برای ساخت درختهای فیلوژنومیک میکروبها. این ابزار هزاران ژنوم مرجع را ادغام میکند و در مطالعات میکروبیوم بسیار کاربرد دارد. -
SILVA’s Phylogenetic Aligner:
ابزاری برای ساخت درختهای مبتنی بر rRNA، با استفاده از پایگاهدادهی معتبر SILVA. -
Nextstrain (برای ویروسها):
یکی از برجستهترین پلتفرمها در فیلوژنی ویروسها است. این ابزار دادههای ژنومی ویروسها (مانند SARS-CoV-2، آنفلوانزا، ابولا) را از پایگاههایی مانند GISAID میگیرد و درختهای زمانمحور (Time-Resolved Phylogenies) میسازد. همچنین تکامل و پراکنش جغرافیایی ویروسها را بهصورت آنلاین و تعاملی نمایش میدهد.
قابلیتهای یکپارچه و چالشها
بسیاری از پلتفرمهای آنلاین از فرمتهای استاندارد مانند FASTA، Newick، Nexus پشتیبانی میکنند. این موضوع امکان ترکیب چندین ابزار در یک جریان کاری را فراهم میسازد. مثلاً:
-
توالیها از GenBank بازیابی شوند،
-
با MAFFT همتراز شوند،
-
درخت با IQ-TREE Web ساخته شود،
-
و سپس در iTOL بصریسازی گردد.
اما باید توجه داشت که روشهای پیچیده در قالب رابطهای ساده میتوانند منجر به سوءبرداشت شوند، اگر کاربر دانش کافی نداشته باشد. برای نمونه، استفاده از تنظیمات پیشفرض بدون بررسی تناسب آنها ممکن است نتایج نادرست ایجاد کند.
۵. مطالعات موردی و کاربردهای ابزارهای آنلاین فیلوژنی
ارزش واقعی ابزارهای آنلاین فیلوژنی زمانی مشخص میشود که در پرسشهای تحقیقاتی عملی در حوزههای مختلف علمی به کار گرفته شوند. هدف اصلی فیلوژنی بازسازی روابط تکاملی است، اما پیامدهای این بازسازیها فراتر از نظریه است.
درختهای فیلوژنتیکی چارچوبی برای سازماندهی تنوع زیستی، تفسیر تنوع ژنتیکی و ارتباط توالیهای مولکولی با زمینههای زیستی، عملکردی یا بالینی فراهم میکنند.
-
در پزشکی، این ابزارها امکان ردیابی پاتوژنها و شناسایی واریانتهای نوظهور را میدهند.
-
در میکروبشناسی، تنوع جوامع میکروبی و مسیرهای تکاملی ژنها را آشکار میکنند.
-
در بومشناسی (Ecology)، الگوهای تنوع زیستی و تعامل گونهها را توضیح میدهند.
-
در طبقهبندی زیستی (Taxonomy)، شواهدی برای دستهبندی سازمانیافتهی موجودات فراهم میکنند.
این بخش به مطالعات موردی و کاربردهای ابزارهای آنلاین فیلوژنی در این حوزهها میپردازد و نشان میدهد که چگونه دسترسی و توانمندی پلتفرمهای وب، تحقیقات بنیادی و کاربردی را دگرگون کرده است.
کاربرد در بیماریهای عفونی و همهگیریها
یکی از جالبترین کاربردها، مطالعه شیوع بیماریهای عفونی است، جایی که تحلیل فیلوژنتیکی در زمان واقعی (Real-time Phylogenetic Analysis) اطلاعات مهمی درباره منشا، انتشار و تکامل پاتوژنها ارائه میدهد.
-
نمونهی واضح، پاندمی COVID-19 است.
-
پلتفرمهایی مانند Nextstrain دادههای ژنومی از GISAID را با ابزارهای بصریسازی قدرتمند ترکیب کردند تا انتشار جهانی SARS-CoV-2 را تقریباً در زمان واقعی ردیابی کنند.
-
پژوهشگران و سیاستگذاران توانستند خطوط ژنی ویروس (Viral Lineages) را شناسایی کرده، ظهور واریانتهای مهم را کنترل کرده و الگوهای فیلوژنتیکی را با دادههای اپیدمیولوژیک (مانند تعداد موارد و توزیع جغرافیایی) مرتبط سازند.
-
درختان تعاملپذیر Nextstrain اجازه دادند تا نه تنها پژوهشگران، بلکه سازمانهای بهداشت عمومی و حتی عموم مردم نیز روند تکامل ویروس را دنبال کنند.
این دسترسی گسترده به اطلاعات فیلوژنتیکی بیسابقه بود و نشان داد که ابزارهای آنلاین چگونه میتوانند فاصله بین تحقیقات علمی و اطلاعرسانی عمومی را کاهش دهند. درسهای آموخته شده از این تجربه احتمالاً آیندهی آمادگی و پاسخ به همهگیریها را شکل میدهند.
کاربرد در مطالعه ژنهای مقاومت به آنتیبیوتیک
در پزشکی، ابزارهای آنلاین برای پیگیری تکامل و انتشار ژنهای مقاومت به آنتیبیوتیک بهکار رفتهاند.
-
مثال: پژوهشگران توزیع جهانی ژنهای Carbapenemase مانند NDM-1 و KPC را بررسی کردهاند.
-
با استفاده از دادههای GenBank و UniProt و ابزارهایی مانند IQ-TREE Web و iTOL، آنها توانستهاند روابط میان سویههای مقاوم و عوامل زمینهای مانند گونه میزبان و محل جغرافیایی را مشاهده کنند.
-
این تحلیلها مکانیزمهای انتقال افقی ژن، نقش پلاسمیدها در انتشار مقاومت و فشارهای تکاملی ناشی از مصرف آنتیبیوتیکها را روشن میکنند.
-
در نهایت، این رویکردهای فیلوژنتیکی به راهبردهای کنترل مقاومت، از جمله برنامههای مدیریت آنتیبیوتیک، پایش هدفمند و توسعه داروهای جدید کمک میکنند.
کاربرد در میکروبشناسی و بومشناسی میکروبی
ابزارهای آنلاین برای تحلیل تنوع گستردهی میکروبی که توسط توالییابی پرسرعت (High-throughput Sequencing) آشکار شدهاند، ضروری هستند.
-
پایگاههای داده rRNA مانند SILVA و Greengenes همراه با ابزارهایی مانند MEGA Online یا PhyML Web به پژوهشگران امکان ساخت درختهایی را میدهند که ساختار جامعه میکروبی را نشان میدهند.
-
ابزارهای بصری مانند iTOL امکان حاشیهنویسی درختها با متادادههایی از شرایط محیطی، میزبان یا ویژگیهای متابولیکی را فراهم میکنند.
-
مثالها:
-
در مطالعات میکروبیوم خاک، درختها نشان دادهاند که خطوط ژنی با تغییرات کاربری زمین، تغییرات اقلیمی یا دسترسی به مواد مغذی چگونه واکنش نشان میدهند.
-
در میکروبیولوژی دریایی، روابط تکاملی میان میکروبهای پلانکتونیک که در چرخههای بیوژئوشیمیایی جهانی نقش دارند، آشکار شدهاند.
-
این ابزارها توالیهای مولکولی خام را به بینشهای اکولوژیک معنیدار تبدیل میکنند.
کاربرد در میکروبیوم انسانی و پزشکی دقیق
-
مطالعات روده انسان با استفاده از درختهای فیلوژنتیکی، ترکیب و روابط تکاملی باکتریها مرتبط با بیماریهایی مانند چاقی، بیماریهای التهابی روده و دیابت نوع ۲ را بررسی کردهاند.
-
ابزارهای آنلاین امکان نقشهبرداری ویژگیهای عملکردی (مثلاً متابولیسم کربوهیدرات یا تولید اسیدهای چرب کوتاه زنجیر) بر روی درختها را فراهم میکنند، و روابط تکاملی را با نتایج بالینی مرتبط میسازند.
-
این رویکرد باعث شناسایی خطوط ژنی محافظتی یا پاتوژنیک و باز شدن مسیرهای درمانی مانند پروبیوتیکها یا پیوند میکروبیوتا شده است.
ابزارهای آنلاین فیلوژنی به طور مستقیم در پیشبرد پزشکی دقیق نقش دارند.
کاربرد در بومشناسی و حفاظت زیستی
-
درختهای فیلوژنتیکی برای ارزیابی تنوع زیستی و تعیین اولویتهای حفاظتی استفاده میشوند.
-
برخلاف معیارهای سنتی تنوع زیستی که بیشتر بر تعداد گونهها تمرکز دارند، رویکردهای فیلوژنتیکی منحصر بودن تکاملی (Evolutionary Distinctiveness) را نیز در نظر میگیرند.
-
مثال: محافظت از گونهای که یک خط تکاملی قدیمی و جداافتاده است، بیشترین کمک به حفظ تاریخچه تکاملی را دارد.
-
ابزارهایی مانند iTOL و PhyML Online برای محاسبه تنوع فیلوژنتیکی در اکوسیستمهای مختلف، از جنگلهای بارانی تا صخرههای مرجانی، استفاده شدهاند.
-
این تحلیلها به شناسایی گونههای EDGE (Evolutionarily Distinct and Globally Endangered) کمک کرده و راهبردهای حفاظتی جهانی را هدایت میکنند.
نقش فیلوژنی در تاکسونومی و سیستماتیک
یکی دیگر از حوزههای کلیدی که ابزارهای آنلاین فیلوژنی در آن تأثیرگذار بودهاند، تاکسونومی و سیستماتیک است. تاکسونومی سنتی عمدتاً بر ویژگیهای مورفولوژیکی تکیه داشت، که گاهی میتواند گمراهکننده باشد به دلیل پدیدههایی مانند تکامل همگرا یا انعطافپذیری فنوتیپی. فیلوژنی مولکولی یک چارچوب عینیتر برای ردهبندی موجودات فراهم میکند و ابزارهای آنلاین به تاکسونومیستها اجازه میدهند فرضیههای مربوط به روابط خویشاوندی را در مجموعههای داده بزرگ آزمایش کنند. برای مثال، ردهبندی مجدد بسیاری از گروههای میکروبی، مانند آرکئاها و برخی شاخههای باکتریایی، با تحلیلهای فیلوژنتیکی توالیهای RNA ریبوزومی ممکن شده است. به همین ترتیب، در گیاهان و حیوانات، فیلوژنی مولکولی باعث بازبینی مرزهای خانوادگی و جنسی شده و اغلب ردهبندیهای دیرینه را به چالش کشیده است.
پلتفرمهای آنلاین، مانع ورود به فیلوژنی را کاهش میدهند و روشهای قابل دسترس برای ساخت درختها و مشاهده روابط فراهم میکنند. ابزارهایی مانند MEGA Online و IQ-TREE Web به تاکسونومیستها اجازه میدهند تحلیلهای دقیق را بدون نیاز به تخصص محاسباتی ویژه انجام دهند و بدین ترتیب مشارکت در تحقیقات سیستماتیک گسترش مییابد.
کاربرد در مطالعه تکامل خانوادههای ژنی
یکی دیگر از کاربردهای جالب، مطالعه تکامل خانوادههای ژنی است. ابزارهای آنلاین به پژوهشگران اجازه میدهند منشأ و تنوع ژنهای خاص را در میان تاکسونها بررسی کنند.
برای مثال، تکامل ژنهای Hox، که نقش حیاتی در توسعه طرح بدن دارند، با استفاده از دادههای بازیابی شده از مخازنی مانند Ensembl و UniProt و تحلیل با پلتفرمهایی مانند PhyML و IQ-TREE مورد بازسازی قرار گرفته است.
درختهای فیلوژنتیکی رویدادهای تکثیر ژنی، واگرایی پارالوگها و نوآوریهای عملکردی را نشان میدهند. چنین مطالعاتی پایههای مولکولی انتقالات تکاملی مهم، از ظهور چندسلولیها تا تنوع مهرهداران، را روشن میکنند. با دسترسپذیر کردن این تحلیلها، ابزارهای آنلاین فیلوژنی به درک عمیقتر از پایههای ژنتیکی نوآوریهای تکاملی کمک میکنند.
کاربرد در علوم کشاورزی
علوم کشاورزی نیز از رویکردهای فیلوژنتیکی که توسط ابزارهای آنلاین تسهیل شدهاند بهرهمند میشوند. برای مثال، درختهای فیلوژنتیکی برای بررسی روابط میان واریتههای محصول، گونههای وحشی مرتبط و آفات استفاده شدهاند. این تحلیلها به پرورشدهندگان کمک میکنند منابع تنوع ژنتیکی را برای افزایش مقاومت محصولات در برابر بیماریها، آفات یا فشارهای محیطی شناسایی کنند.
پلتفرمهای فیلوژنتیکی آنلاین به پژوهشگران کشاورزی اجازه میدهند مجموعه دادههای ژنومی بزرگ را یکپارچه کنند، درخت بسازند و روابط را به شکلی بصری نمایش دهند که استراتژیهای عملی پرورش محصول را هدایت کند. برای مثال، فیلوژنی واریتههای برنج و گندم راهنمای انتقال صفات مفید از گونههای وحشی به ارقام زراعی بوده است. به همین ترتیب، مطالعات فیلوژنتیکی آفات حشرهای و پاتوژنهای قارچی بینشهایی درباره منشاء شیوعها و مکانیزمهای تطبیق میزبان ارائه کردهاند. در هر مورد، ابزارهای آنلاین فیلوژنی پل ارتباطی میان زیستشناسی تکاملی پایه و نوآوریهای کاربردی کشاورزی فراهم میکنند.
کاربرد در علوم قانونی و جرمشناسی
فیلوژنی همچنین در علوم قانونی و کاربردهای قضایی نقش دارد. ابزارهای آنلاین در مواردی که شواهد ژنتیکی باید در چارچوب تکاملی قرار گیرد، مورد استفاده قرار گرفتهاند. برای مثال، تحلیلهای فیلوژنتیکی در پروندههای انتقال HIV استفاده شدهاند، جایی که تشخیص اینکه آیا سویههای ویروسی افراد مختلف به هم نزدیک هستند یا نه، میتواند پیامدهای قانونی داشته باشد.
با اینکه چنین کاربردهایی نیازمند تفسیر دقیق و رعایت استانداردهای روششناسی هستند، اما نشان میدهند که تحلیلهای فیلوژنتیکی دامنه اثر بسیار وسیعی دارند. دسترسپذیری ابزارهای آنلاین تضمین میکند که این تحلیلها با شفافیت و قابلیت بازتولید انجام شوند، که در زمینههای قانونی اهمیت بالایی دارد.